薛愿超课题组发展了全景式捕获RNA空间互作的研究方法RIC-seq
5月21日,中国科学院生物物理研究所薛愿超课题组在Nature Protocols杂志上在线发表了题为“Global in situ profiling of RNA-RNA spatial interactions with RIC-seq”的研究论文。
为了捕获细胞内RNA原位高级结构和非编码RNA作用靶标,薛愿超课题组先前开发了RIC-seq(RNA in situ conformation sequencing)新技术(Cai et al., Nature, 2020)。该方法利用甲醛对细胞进行交联以固定RNA-RNA之间的相互作用,在保持细胞完整性的前提下通过RNA原位近端连接技术将空间邻近的RNA片段连接起来形成嵌合体RNA,并创新性地使用pCp-biotin标记和富集嵌合体RNA,能够以更低的假阳性率和测序成本实现单碱基分辨率下全景式解析细胞内各种编码及非编码RNA的空间相互作用。RIC-seq方法在Nature杂志发表后,受到了Nature Methods期刊的亮点评论,并获得了Nature Reviews Cancer、Nature Reviews in Molecular Cell Biology、JCI等权威杂志综述推介和引用。
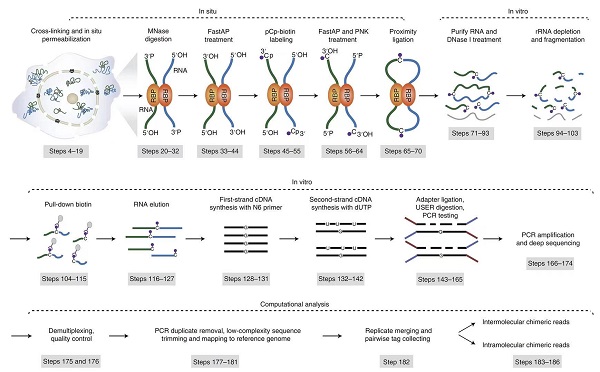
RIC-seq建库和分析流程图
为了方便科研工作者使用RIC-seq技术并拓展其应用范围,薛愿超课题组在本论文中详细描述了RIC-seq技术的设计原理、开发过程、优缺点、实验步骤及时间、预期结果和问题诊断等。在本文中,薛愿超课题组展望了RIC-seq的应用场景,主要包括:RNA二级与原位高级结构重建,非编码RNA作用靶标鉴定,增强子-启动子调控网络构建,疾病相关非编码突变致病机理解析,RNA病毒基因组空间构象模拟以及RNA药物开发等。
该项工作得到了科技部重点研发计划、国家自然科学基金委和中科院先导项目等多项基金资助。生物物理所副研究员曹唱唱、博士后蔡兆奎为论文共同第一作者,薛愿超研究员为论文通讯作者。